
Por: Jaime Aguilar y Michael Hernández Segura.
Chrystian Camilo Sosa Arango, es biólogo egresado de la Universidad del Quindío y actualmente está terminando el doctorado en Ingeniería y Ciencias Aplicadas de la Pontificia Universidad Javeriana Cali. Tiene experiencia en informática de la biodiversidad, manejo de grandes volúmenes de datos, análisis espaciales y metagenómica. Ha trabajado en organizaciones aliadas como el CIAT, en la administración de información espacial de parientes silvestres de cultivos y en otros aspectos asociados con manejo de datos en el agro, como la asociación de la deforestación y conflicto armado.
En el Programa ÓMICAS, trabaja en el análisis de datos biológicos dentro del proyecto de Genómica, conjuntamente con los profesores Mauricio A. Quimbaya de #JaverianaCali y Victor Hugo García-Merchan de la Universidad del Quindío. De manera particular, su interés dentro del proyecto es la identificación de genes y circuitos metabólicos para la tolerancia del Aluminio en las plantas de arroz.
El impacto del Aluminio como de otros metales pesados en una planta, es que entra en competencia con los nutrientes que absorbe de su entorno, afectando su crecimiento y por tanto su productividad. La investigación de Chrystian se basa en el análisis metabólico en arroz y de genómica comparativa con la especie Arabidopsis thaliana (referencia de estudio), expuesta a estrés por este metal pesado.
Estos experimentos de enriquecimiento funcional se orientan al mejoramiento de la capacidad antioxidante de la planta cuando se somete a situaciones de estrés. Y lo que se busca es que los cultivos de arroz sean más tolerantes al estrés por aluminio. En su investigación, analiza diferentes variedades de arroz para la identificación de genes y sus circuitos metabólicos en aquellas que son susceptibles y tolerantes al estrés por Aluminio.
En su trabajo, Crhystian realiza procesos de simulación de genes candidatos para el análisis y realiza una reconstrucción metabólica a escala genómica, esto es que, a nivel computacional identifica qué proteínas y rutas están actuando y cómo esto favorece o inhibe el crecimiento de raíz en arroz. Dentro de sus resultados, ha encontrado que los genes del arroz están asociados a mecanismos de exclusión y unas estrategias metabólicas propias ante el estrés del aluminio, representando un avance significativo en la genómica comparativa al identificar elementos conservados entre plantas muy alejadas genéticamente y creando un nuevo software que puede ser usado fácilmente por biólogos para realizar análisis comparativos funcionales.
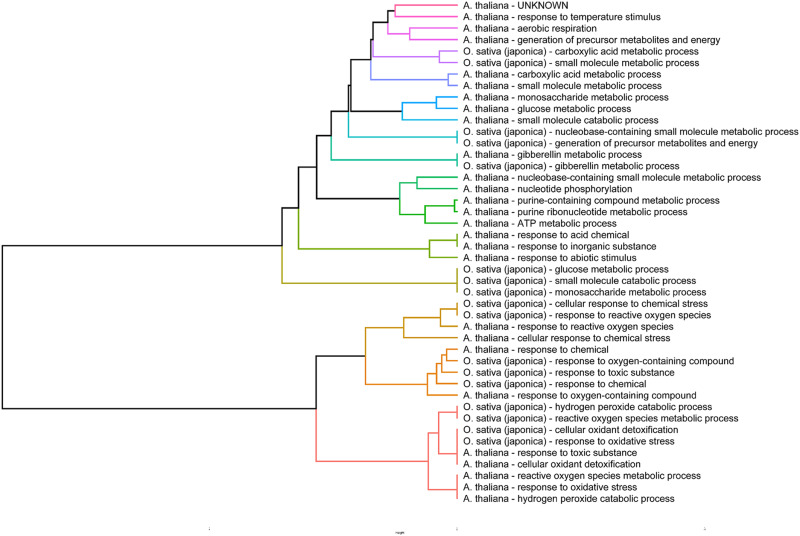
Estos avances desde el Programa ÓMICAS, han contribuido en su capacidad investigativa de comparar especies a través del software GOCompare, fortaleciendo desde ese enfoque su experiencia en el área de la bioinformática.
Adicionalmente, Chrystian realizó su estancia doctoral en una de las universidades aliadas de la alianza, la Universidad de Gante (Ghent University) en Bélgica, particularmente en el VIB-UGent Center for Plant Systems Biology. Durante su estancia, trabajó con herramientas asociadas a la transcriptómica para la identificación de genes candidatos a tolerancia de estrés a través de análisis bioinformáticos y estrategias de aprendizaje de máquina (machine learning), bajo la tutela del profesor Klaas Vandepoele.
El objetivo de su pasantía fue encontrar genes diferencialmente expresados asociados a sequía en trigo y los genes regulando estos. Para dicho fin, realizó una búsqueda de estudios con datos transcriptómicos presentes en donde hubiesen plantas de trigo en condiciones de sequía y como control, plantas con irrigación normal para construir un atlas transcriptómico de diversos estudios donde se reflejase trigo en diversas condiciones de sequía.
De esta manera, utilizó herramientas como nf-core/rnaseq y desarrolló una nueva herramienta bioinformática escrita en R para encontrar genes diferencialmente expresados a través de la librería edgeR para encontrar genes pan-sequía (que al menos se encuentran en alguna condición de sequía contra control) y genes core (que siempre estuviesen expresándose).
A su vez, se usó un nuevo algoritmo llamado vHRR el cual permite predecir nuevas funcionalidades de genes a través de aprendizaje automático y finalmente, a través de enriquecimiento funcional, encontrar motivos (porciones pequeñas de ADN las cuales ayudan a identificar genes que regulan la transcripción de estos genes principales asociados a sequía). Como resultado de esta investigación, se identificaron 2.8431 y 261 genes pan-sequía y core, así como 3.538 y 438 nuevos genes candidatos que no se habían reportado con sequía previamente. Finalmente, miembros de las familias de factores de transcripción: AP2, bZIP, and Myb/SANT, bHLH, E2F, HSF, AT hook, Dof, NAC/NAM, y NAC/NA fueron fuertemente enriquecidas demostrando su rol para la regulación de este estrés en trigo.
Las nuevas herramientas bioinformáticas creadas en esta experiencia servirán para estudios futuros en arroz y caña de azúcar con el fin de obtener una mejor visión a nivel transcriptómico con relación a estreses abióticos como el de iones de aluminio o sequía.
El aporte del trabajo de Crhystian a los grandes objetivos del programa está en la Seguridad Alimentaria, puesto que su aporte investigativo está orientado al mejoramiento genético de las plantas de arroz para generar nuevas variedades, siendo este cultivo, uno de las principales fuentes de carbohidratos en la alimentación de Latinoamérica.
Conozca más sobre este trabajo en: https://www.sciencedirect.com/science/article/pii/S0888754322002737


















